【学术成果】邓柯课题组博士生提出全新预测模型,提高microRNA调控网络预测精度

近日,清华大学统计学研究中心2017级博士生李祺与中国医学科学院肿瘤医院王攀博士以共同第一作者身份撰写的论文“MiRACLe: an individual-specific approach to improve microRNA-target prediction based on a random contact model”被Briefings in Bioinformatics接收并在线发表。Briefings in Bioinformatics影响因子8.990,是计算生物学和生物医学方法研究领域的国际顶尖期刊。我中心邓柯副教授与中国医学科学院肿瘤医院赫捷院士作为论文的共同通讯作者,联合指导了相关研究和论文撰写。
该论文提出了一种基于随机碰撞模型的miRNA靶点预测模型miRACLe,提高了microRNA(miRNA)的靶点识别的精确度和准确度,对于诊断和治疗具有重要的意义。miRNA的调控具有高度样本特异性,目前现有的模型对于特异性样本的计算准确度以及对诊断的辅助作用有限。本论文中的模型将多种miRNA和mRNA的序列信息以及表达谱信息进行整合,可以实现对单细胞或单样本表达谱的miRNA调控靶点预测,下图是该模型的计算流程图,其中矩阵![]() 即为该算法计算得到的miRNA-mRNA之间的miRACLe得分,基于此得分即可得到不同miRNA的靶点预测结果。
即为该算法计算得到的miRNA-mRNA之间的miRACLe得分,基于此得分即可得到不同miRNA的靶点预测结果。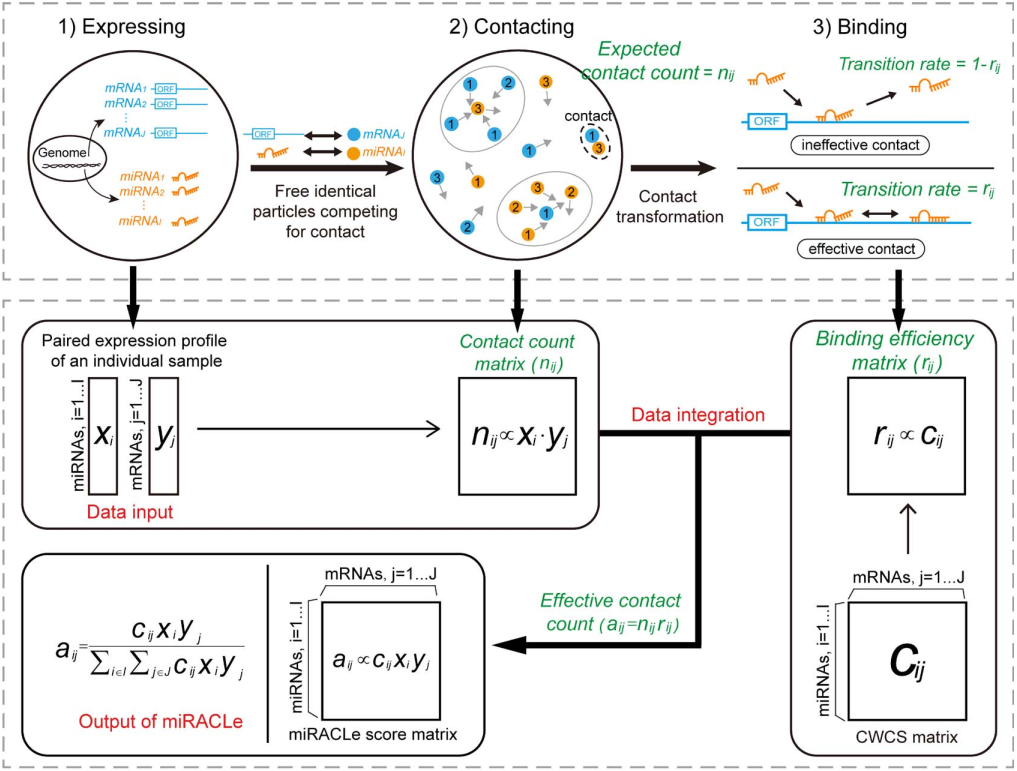
通过在多个数据集上对miRACLe和已有算法进行比较,论文发现该算法在预测精度、miRNA转染实验以及肿瘤相关基因的富集性分析等多个维度上均较已有方法有更好的预测效果,并且具有更快的计算速度。值得注意的是,miRACLe算法在多种不同的生物学背景,多种不同类型的表达谱以及多个验证数据集上的表现均一致优于已有方法。
最后,该论文还尝试将miRACLe的模型框架应用到其他多种基于序列信息的方法上,例如DIANA microT-CDS、miRanda-mirSVR和MirTarget4。结果表明通过使用miRACLe的算法框架与表达谱信息相结合,可以提高其预测精度和其他生物学表现,这也进一步拓展了该算法的应用范围。
论文网址:
