近日,2022年第60届国际计算语言学协会年会(Annual Meeting of the Association for Computational Linguistics,简称ACL)举行,我中心邓柯课题组18级博士研究生潘长在、俞声课题组17级博士研究生袁正、18级博士研究生罗声旋几位同学的多篇投稿文章被接收。ACL会议始于1962年,由国际计算语言学协会主办,是自然语言处理与计算语言学领域最高级别的学术会议。
潘长在同学的论文入选“主会长文”单元,题为“ TopWORDS-Seg:开放域中文文本领域通过贝叶斯推断同时进行文本切词和词语发现的方法 (TopWORDS-Seg: Simultaneous Text Segmentation and Word Discovery for Open-Domain Chinese Texts via Bayesian Inference)”,文章针对于几十年来计算语言学中的一个关键瓶颈,开放域中文文本处理问题展开论述。称之为瓶颈是因为在开放域这种具有挑战性的场景中,文本分词和词语发现经常相互纠缠,且并无可用的训练数据。尚无现有方法可以在开放域中同时实现有效的文本分词和单词发现。该文章通过提出一种基于贝叶斯推理的名为 TopWORDS-Seg 的新方法来填补这一空白,在没有训练语料库和领域词表的情况下具有很好的表现和解释性。该文章通过维基百科数据用一系列实验研究证明了 TopWORDS-Seg 的优势。潘长在是第一作者,邓柯副教授作为通讯作者与清华大学计算机系科学与技术系的孙茂松教授共同指导了该工作。
袁正同学共有三篇文章入选:
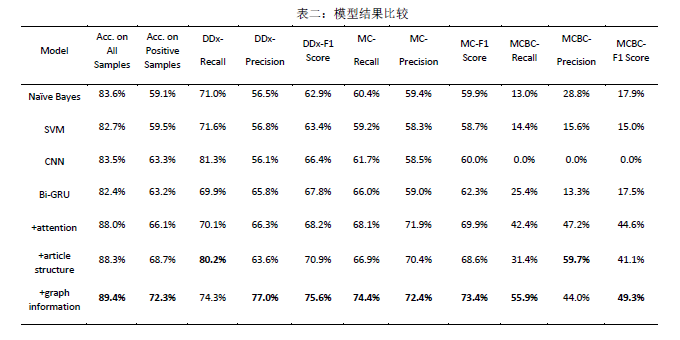
入选“主会短文”单元文章:“基于疾病同义词的匹配网络的自动疾病编码(Code Synonyms Do Matter: Multiple Synonyms Matching Network for Automatic ICD Coding)”通过额外利用疾病编码的同义词信息去匹配电子病历中的不同文本以达到更好的自动疾病编码效果,在MIMIC-3电子病历数据集上得到了超过以往方法的分类效果。
入选“Findings长文”单元文章 :“使用三仿射融合异质信息的嵌套命名实体识别方法(Fusing Heterogeneous Factors with Triaffine Mechanism for Nested Named Entity Recognition)”通过三仿射变换改进基于片段分类的命名实体识别模型中的片段表示和片段分类方法,在新闻和医疗命名实体识别任务上取得了超过之前的结果。以上两篇文章袁正均为第一作者,与阿里巴巴达摩院团队合作完成。
此外,袁正与浙江大学、鹏程实验室等研究团队合作的论文:“中文医学自然语言处理评测数据集(CBLUE: A Chinese Biomedical Language Understanding Evaluation Benchmark)也入选了“主会长文”单元。
罗声旋同学的论文入选“Findings长文”单元,题为“联合实体对齐和悬空实体识别的高精度无监督方法 (An Accurate Unsupervised Method for Joint Entity Alignment and Dangling Entity Detection) ”,罗声旋为该文的第一作者,其导师俞声副教授为通讯作者。文章针对在对齐两个知识图谱的现实场景中的三个主要问题:(1)不存在等价对应的实体,也即悬空实体,广泛存在于知识图谱中;(2)悬空实体标签和实体对(等价的两个实体)标签难以获得,一个普适的知识图谱对齐方法需要尽可能避免对监督数据的依赖;(3)各对齐之间以及预测对齐与识别悬空实体之间是互相影响的,需要整体地考虑识别悬空实体并对齐等价的实体。该文章首先根据实体的文本语义信息和全局的相似性指导两个知识图谱中的实体嵌入的训练,从而获得实体之间的距离估计。然后,给每个知识图谱添加一个虚拟实体,从而把实体对齐和悬空实体整合为一个统一的最优运输问题,并解这个问题。最终,与虚拟实体对齐的实体为悬空实体,其余对齐为模型预测的等价实体对。一系列实验表明,该文章在不依赖监督数据的情况下,能够达到当前实体对齐任务上的最优表现,并且有高质量的悬空实体识别结果。


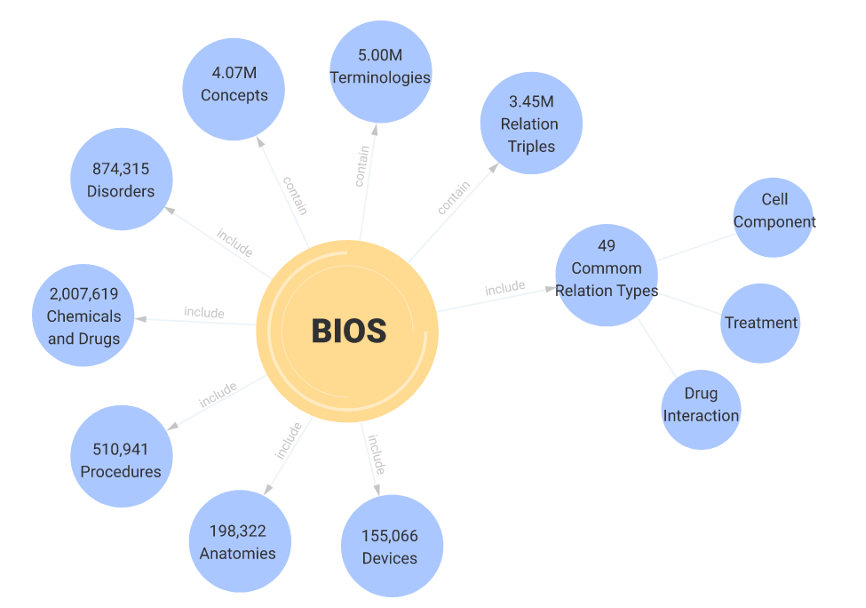
 11月22-23日,深圳市福田区人民政府、深圳市福田区科技创新局和粤港澳大湾区数字经济研究院(International Digital Economy Academy,简称“IDEA”)联合举办IDEA大会。IDEA创院理事长、美国国家工程院外籍院士、英国皇家工程院外籍院士沈向洋在会上发布了由清华大学统计学研究中心俞声副教授团队与粤港澳大湾区数字经济研究院联合开发的大型开放医学知识图谱(Biomedical Informatics Ontology System,简称“BIOS”)(http://bios.idea.edu.cn)。
11月22-23日,深圳市福田区人民政府、深圳市福田区科技创新局和粤港澳大湾区数字经济研究院(International Digital Economy Academy,简称“IDEA”)联合举办IDEA大会。IDEA创院理事长、美国国家工程院外籍院士、英国皇家工程院外籍院士沈向洋在会上发布了由清华大学统计学研究中心俞声副教授团队与粤港澳大湾区数字经济研究院联合开发的大型开放医学知识图谱(Biomedical Informatics Ontology System,简称“BIOS”)(http://bios.idea.edu.cn)。


 近日,统计学研究中心2016级博士生徐嘉泽与清华大学自动化系江瑞教授团队、斯坦福大学统计系Wing Hung Wong教授团队的合作文章“Density estimation using deep generative neural networks”在《美国科学院院刊》(PNAS)在线发表。徐嘉泽同学于2020年1月赴美国斯坦福大学进行为期近一年的访问学习,在访问期间主要参与了Wing Hung Wong教授实验室的文本分析、贝叶斯蒙特卡洛算法开发等方面的工作。在本项目中,徐嘉泽同学参与了模型构建和工具开发等工作。
近日,统计学研究中心2016级博士生徐嘉泽与清华大学自动化系江瑞教授团队、斯坦福大学统计系Wing Hung Wong教授团队的合作文章“Density estimation using deep generative neural networks”在《美国科学院院刊》(PNAS)在线发表。徐嘉泽同学于2020年1月赴美国斯坦福大学进行为期近一年的访问学习,在访问期间主要参与了Wing Hung Wong教授实验室的文本分析、贝叶斯蒙特卡洛算法开发等方面的工作。在本项目中,徐嘉泽同学参与了模型构建和工具开发等工作。